2.中国林科院林业研究所细胞生物学实验室, 北京, 100091
 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2011 年, 第 9 卷, 第 26 篇 doi: 10.5376/mpb.cn.2011.09.0026
收稿日期: 2011年01月18日 接受日期: 2011年02月28日 发表日期: 2011年03月07日
栗婷等, 2011, 落叶松体细胞胚胎全长cDNA文库构建及初步分析, 分子植物育种 Vol.9 No.26 (doi: 10.5376/mpb.cn.2011.09.0026)
以日本落叶松胚性细胞系638#体细胞胚胎发育多个时期的胚性组织为材料,利用SMART技术构建了落叶松体细胞胚胎全长cDNA文库。经检测,所构建初始文库滴度为2.25×106 pfu/mL,重组率为95.13%,扩增文库滴度为2.65×109 pfu/mL,插入片段长度主要分布在0.5~2 kb之间,平均长度为850 bp;所构建文库能够用于目的基因分离筛选及表达。从初始文库中随机挑选16个克隆进行测序,成功测序15个,经去除低质量序列并拼接,获得13个unigenes,包括 2个 contigs 和11个 singletons,其中3个为已知功能 unigenes,2个 unigenes 具有推测功能,2个 unigenes 功能未知,另外6个 unigenes 在蛋白质数据库中无同源序列,所构建的文库为从分子水平上揭示落叶松体细胞胚胎发育机理,进一步发现与诠释关键基因功能及其表达奠定物质基础。
植物体细胞胚胎发生体系作为细胞全能性表达的重要途径,在人工种子、种质保存、无性繁殖、遗传转化等诸多领域具有重要应用价值(张蕾等, 2009; 吕守芳等, 2003),也为胚胎发育过程中各种生物学事件研究提供理想的模式体系。迄今,已有胡萝卜(Steward et al., 1958)、拟南芥(Ikeda-Iwai et al., 2003)、小麦(Cuming and Lane, 1979)、水稻(Litts et al., 1992)、玉米(Williams and Tsang, 1991)、大豆(王萍等, 2004)、棉花(Davidonis and Hamilton, 1983)等多种植物通过体细胞胚胎发生获得再生植株;在林木上,国内外已从冷杉属(Abies)、落叶松属(Larix)、云杉属(Picea)松属(Pinus)等多种针叶树中获得体细胞胚。落叶松作为重要针叶造林用材树种,在体细胞胚胎发生及再生体系方面已经取得重要突破(吕守芳等, 2005),但与很多被子植物相比,其体细胞胚胎发生存在质量不高、发生数量低,且难以同步化等关键技术问题,成为落叶松规模化无性繁殖及遗传转化的瓶颈(吕守芳等, 2004, 林业科学研究, 17(3): 392-404)。亟需从植物体细胞胚胎发生的机理及调控方面开展研究;目前,已分离与鉴定了多种与体细胞胚发生相关的基因(张蕾等, 2006; Pullman and Webb, 1994; Palovaara et al., 2010)。但从体细胞诱导成胚性细胞、再由胚性细胞发育成体细胞胚是一个复杂的过程,迄今,对植物体细胞胚胎分子机理的研究相对薄弱,尤其是针叶树体细胞胚机理研究极少。
利用现代功能基因组学手段是开展分子生物学研究的重要方式。构建全长cDNA文库可以高效、大规模获得基因序列信息,对基因组庞大,近期内尚不能进行全基因组测序的物种来说,是进行其功能基因组研究的一条有效途径(毛新国等, 2006)。目前,已有多种构建全长cDNA文库的方法,其中主要有:CAPture法、Oligo-capping法、SMART (Switching Mechanism At 5,end of RNA Transcript method/SMART method)法、Cap-jumping法以及Cap-trapper法等。SMART (Zhu et al., 2001; http://www.clontech.com/images/PT3000-1.pdf)技术是Clontech公司的一项专利,是目前国内应用最多、较为成熟的一种全长cDNA文库构建技术,和目前其它市场化的全长cDNA文库构建技术相比,有其独特的优点:SMARTScribeTM MMLV逆转录酶的末端转移酶活性以及双链cDNA的分级分离保证了连接片段的全长率,此外,利用此方法构建文库时,所需起始材料用量少(0.025-1.0 μg的mRNA或者0.05-2.0 μg的总RNA)且可实现定向克隆。该方法快速、操作简单、经济。综合以上因素,本研究选用SMART技术构建落叶松体细胞胚胎全长cDNA文库。
本研究首次以日本落叶松(Larix leptolepis)胚性细胞系638#原胚团和早期单胚为实验材料,利用SMART技术,构建落叶松体细胞胚胎全长cDNA文库,进一步揭示落叶松体细胞胚胎发育分子机理,为探索基因功能、表达谱研究奠定基础。也为研究合子胚发生机理提供科学依据。
1 结果与分析
1.1 总RNA及mRNA的质量
RNA质量的高低直接影响到文库的质量,提取的总RNA经1%琼脂糖电泳检测,显示明显的28S、18S、条带,28S、18S条带亮度比约为2:1,表明RNA完整(图1);ND-1000检测得A260/A280比值均在2.1左右,A260/A230比值均在1.8以上(表1),说明RNA纯度高,无DNA、蛋白质及小分子的污染;从各总RNA中取出2 μL,于37℃温浴2h以上,电泳检测后与原RNA无明显区别,说明没有RNase的污染(图2)。纯化后的mRNA1%琼脂糖电泳检测结果显示获得的mRNA呈均匀弥散状,表明mRNA无降解,质量好。
.png) 图1 各时期材料总RNA电泳图 Figure 1 Electrophoresis of total RNA from three development stage of embryogenic callus |
.png) 图2 温浴处理后RNA电泳图 注: M: 1kb DNA Marker; 1、2、3: 原各时期材料总RNA; a、b、c: 温浴处理后各时期总RNA图 Figure 2 Electrophoresis of total RNA after incubation Note: M: 1kb DNA Marker; 1、2、3: the primary total RNA from the three development stage of Embryonic tissue; a、b、c: the total RNA after incubation from three development stage of Embryonic tissue |
|
表1 总RNA NanoDrop-1000检测结果 Table 1 NanoDrop-1000 analysis of total RNA |
1.2 mRNA合成双链cDNA
取5 μL合成的双链cDNA于0.8%的琼脂糖凝胶电泳检测,双链cDNA呈弥散状,弥散范围重点分布在0.5kb-2kb之间(图3),Nano-Drop1000检测得,A260/A280比值为1.86,A260/A230比值为在1.84,浓度为546.6 ng•μL-1。这表明双链cDNA条带正常且量足,满足建库要求。
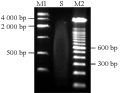 图3 mRNA反转录成双链cDNA电泳图 注: M1: 1kb DNA Marker; S: 样品双链cDNA; M2: 100bp DNA marker Figure 3 Electrophoresis of dscDNA Note: M1: 1kb DNA Marker; S: dscDNA; M2: 100bp DNA marker |
1.3 分级分离后双链cDNA
分级分离后双链cDNA各管产物于1.1%的琼脂糖凝胶电泳检测(图4),表明:1-6管几乎无DNA片段;从第七泳道开始出现cDNA片段,且cDNA长度逐渐变小;7-10的片段基本大于500 bp;11以后各管有小于500 bp的片段,为避免过多的小片段与载体连接,保证文库的全长率,舍弃10以后的cDNA产物,收集合并7、8、9、10这4产物。用于后续实验。
|
图4 分级分离后cDNA电泳图 注: M: 1kb DNA mareker; 1-15: 分级分离后各管cDNA Figure 4 Electrophoresis of dscDNA after size fractionation Note: M: 1kb DNA mareker; 1-15: represent the dscDNA after size fractionation |
1.4 cDNA文库质量鉴定
在3个cDNA与λTriplEx2载体连接反应中,根据计算所得文库滴度来判断,体系A连接效果最佳。
cDNA与载体的比例为0.5:1,对各稀释度噬菌斑数计数后,按照文库滴度公式计算得到初始文库滴度为2.25×106 pfu/mL(图5),扩增文库滴度为2.65×109 pfu/mL。蓝白斑计数测定得到文库重组率为95.13%。
随机挑取22个噬菌斑进行PCR扩增鉴定,估计文库的插入片段大小。电泳结果(图6)显示,其中一条未有特异条带,一条片段约在200 bp左右,根据载体信息判断其可能为空载体,其它片段均在500 bp以上,分布在0.5~4 kb之间,平均长度约0.85 kb。
 图5 初始文库滴度测定 Figure 5 Titer the primary library |
|
图6 文库插入片段的随机PCR检测 注: M: 1 kb DNA Marker; CK: 阴性对照(无DNA模板); 1-22: 菌落PCR片段 Figure 6 PCR insert screening of the primary library Note: M: 1 kb DNA Marker; CK: negative control(no DNA template); 1-22: colony PCR products |
1.5 ESTs的分析
随机选取初始文库中16个克隆转化为质粒后测序,获得有效序列15个,测序成功率为94.0%。经序列拼接后得到13条Unigenes,其中2个Contigs。其中碱基最长的Unigene达1077bp。拼接后获得的Unigenes与GenBank中的蛋白质数据库比对,发现其中具有已知功能Unigenes3个,具有推测功能Unigenes2个,未命名或未知的功能Unigenes2个,在蛋白质数据库中无同源序列的功能未知Unigenes6个(表2)
 表2 cDNA克隆有效序列比对结果 Table 2 Result of comparison of effective sequences of cDNA clones |
2 讨论
2.1 全长cDNA文库构建
总RNA质量直接影响到文库质量,而影响RNA质量的因素主要有三个:一是否有降解;二是否有污染(DNA、蛋白质、小分子物质);三是否完整(龙松华等, 2008)。RNA酶的大量存在是导致RNA降解的主要原因,本研究中提取RNA时采取以下措施大大地降低了RNA降解的几率:采用固相Rnase清除剂(Surface RNase Erasol)擦拭试验台面、移液器、镊子等无法通过高温或者灭菌处理的器材;试验过程中及时更换手套;在超净工作台中提取RNA。落叶松胚性组织中多糖、多酚含量较高,为了减少糖和酚类物质的污染,挑取材料时尽量选择新长出的、健康的表层的胚性组织。并且严格按照Total RNA Purification Kit和OligotexTM mRNA Purification Kit说明书提取总RNA和分离mRNA,保证得到高质量、高纯度的总RNA和mRNA。构建cDNA文库时,以体细胞胚发育三个时期的胚性组织为材料,分别进行取样,再把所提取的总RNA等量混合,以保证体细胞胚发育相关RNA种类的完整性。
文库的滴度、重组率及插入片段的大小是鉴定cDNA 文库质量的重要指标(周祥明等, 2007)。当要求克隆到某低丰度mRNA的概率为99%时,文库的克隆数不应低于1.7×105个。文库的重组百分率应达到80%以上(http://www.clontech.com/images/PT3000-1.pdf),本文库滴度为2.25×106 pfu/mL,重组率为95.13%,说明构建的落叶松体细胞胚胎cDNA文库满足构建高质量文库的要求,文库的代表性较高一般认为,非哺乳动物如植物、昆虫、酵母等的PolyA+RNA分布在0.5~3.0 kb之间(周祥明等, 2007)。本实验中双链cDNA的凝胶电泳图显示cDNA分布在0.3-4 kb之间,对随机挑取的22个克隆进行PCR扩增检测,观察到扩增片段范围为0.5-4 kb之间,这说明库内的cDNA序列具有完整性。
2.2 部分克隆序列分析
在成功构建落叶松体细胞胚胚胎全长cDNA文库的基础上,对少数部分序列进行随机克隆和ESTs测序,并对测序结果做了简单分析,但13条Unigenes中有6条序列不能在蛋白质数据库中找出其同源序列。经分析可能有以下原因:首先蛋白质数据库有限,一些新基因在数据库中不存在同源蛋白质(王博等, 2008);其次,研究表明,针叶树种胚胎发育的分子生物调控机理和目前研究较为深入的拟南芥还是有很大差异(Cairly and Pullman, 2007)。由于缺少参考基因组信息,对针叶树功能和比较基因组学的研究受到限制,且针叶树胚胎与其它组织在基因表达上也有显著不同(Ralph et al., 2008),这都可能导致本研究中较多体细胞胚胎的cDNA序列在数据库无同源序列;再次可能是目前数据信息挖掘不够,BLASTX的依据是氨基酸序列相似性,而氨基酸序列差异较大的蛋白可能会具有相同功能(王博等, 2008)。随着蛋白质数据库在不断增大、更新,将来一些未知功能基因会得到注释。
落叶松体细胞胚胎全长cDNA文库的构建,为探索落叶松体细胞胚胎发育过程奠定基础,也可为研究其它树种体细胞胚胎发育、合子胚发育提供科学依据,本实验室欲从库中克隆与调控落叶松体细胞胚胎发育相关基因、microRNA靶基因等,进一步揭示落叶松体细胞胚胎发育过程及其调控机理。
3 材料与方法
3.1 实验材料
松杉类等针叶树体细胞胚胎发育过程分为三个阶段:原胚团时期(proembryogenic mass)、胚胎形成早期(early embryogeny)和胚胎形成晚期(late embryogeny) (Filonova et al., 2000)。为了最大程度获得落叶松体细胞胚胎发育相关基因,以落叶松胚性细胞系638#继代培养48 h、15 d以及体细胞胚成熟阶段48 h的胚性组织为供试材料(吕守芳等, 2005)。各材料取材后于液氮中保存备用。
3.2 研究方法
3.2.1 试剂
Total RNA Purification Kit购自Norgen公司,OligotexTM mRNA Purification Kit购自Qiagen公司,SMART cDNA Library Construction Kit和Advantage 2 PCR Kit购自Clonetech公司,MaxPlaxTM Lambda Packaging Extracts噬菌体包装蛋白购自Epicenter公司。DL10,000 DNA Marker购自TaKaRa公司。Surface Rnase Erasol (固相RNase清除剂)购自北京天恩泽基因科技有限公司。
3.2.2 总RNA的提取及质量鉴定
取50 mg各材料在液氮中快速研磨成粉末状,然后依照Total RNA Purification kit试剂盒提取总RNA,用ND-1000测定总RNA的浓度和纯度,1%琼脂糖电泳检测RNA完整度;取2 μL各时期材料总RNA,37℃温浴2 h以上,1%琼脂糖电泳检测,与原总RNA比较,判断有无RNase污染。
3.2.3 mRNA的分离
将上述3个时期材料总RNA等量混合,采用OligotexTM mRNA Purification Kit,从总RNA中分离mRNA,ND-1000测定浓度及纯度,1%琼脂糖电泳检查mRNA质量。
3.2.4 双链cDNA的合成
取3 μL纯化的mRNA(约36 ng),加入1 μL SMART IV Oligo nucleotide (10 μmol•L-1)、1 μL CDSIII/3,PCR Primer (10 μmol•L-1)短暂离心混匀,72℃ 2 min后置于冰上放置2 min,再依次加入2 μL的5×第一链反应缓冲液、1 μL DTT (20 mmol)、1 μL dNTP Mix (10 mmol)、1 μL SMARTScribeTM MMLV Reverse Transcriptase参照SMARTTM cDNA Library Construction Kit说明,合成cDNA第一链。PCR反应条件:95℃ 20 sec,31个循环:95℃ 5 sec,68℃ 6 min;4℃结束反应,反应体系为50 μL。
引物及寡核苷酸片段序列如下:
SMART IV Oligo nucleotide
5’-AAGCAGTGGTATCAACGCAGAGTGGCCATTATGGCCGGG-3’;
CDSIII/3’PCR Primer
5’-ATTCTAGAGGCCGAGGCGGCCGACATGd(T)30 N一1N-3’;
5’PCR Primer
5’-AAGCAGTGGTATCAACGCAGAGT-3’
3.2.5 cDNA分级分离
合成的双链cDNA经蛋白酶K消化和SfiI酶切后过CHROMA SPIN-400柱子进行分级分离,共收集15管,每管取3 μL进行1%琼脂糖电泳检测,根据电泳结果,取大于500 bp的片段混合并按照SMART cDNA Library Construction Kit说明浓缩,溶于7μL去离子水中。
3.2.6 cDNA与载体的连接及包装
cDNA与λTriplEx2载体的连接设3个连接梯度(表3),反应体系为5 μL,16℃下连接过夜。然后按照MaxPlaxTM Lambda Packaging Extracts方法包装。用500 μL phage dilution Buffer稀释后再加入25 μL氯仿。轻轻涡旋混匀,于4℃储藏。
|
表3 cDNA与载体连接反应的体系 Table 3 Reaction systems of ligating cDNA to vector |
3.2.7 文库质量检测及扩增
将包装得到的cDNA文库按10-2、10-3、10-4、10-6进行稀释,每梯度设置3个重复,取100 μL的稀释液和100 μL OD值约为0.8-1.0的XL1-Blue混合,37℃吸附15 min。再与2 mL上层琼脂混匀,倒在预热的LB平板上,37℃培养6-18 h,待噬菌体长出后,计数噬菌斑,按照公式:文库滴度=平板上克隆数×稀释倍数×1000/涂板体积(μL)计算cDNA文库滴度。根据cDNA文库滴度,按照文库构建试剂盒说明,在顶层琼脂中加入IPTG和X-gal,通过蓝白斑计数进行重组率的测定。从初始文库中随机挑取22个噬菌斑,加入5′端(5′PCR CTCGGGAAGCGCGCCATTCTCTTGG)和3′端(3′ PCR ATACGACTCACTATAGGGCGAATTGGCC)引物′进行PCR鉴定,确定文库平均插入片段。
文库的扩增和保存按照Clontech试剂盒说明书的方法进行,取适量扩增后的文库以10-2、10-4、10-6、10-8的稀释度测定扩增文库滴度,每个稀释度设置3个重复。待噬菌斑长出后计数,计算扩增文库滴度。
3.2.8 文库序列测定及分析
从初始文库中随机挑取16个噬菌体克隆,为操作方便,先将这16个克隆分别转化为质粒克隆:将各阳性克隆接种到350 μL的1×Lambda dilution Buffer。涡旋混匀,37℃,200-250 rpm下培养3-4 h后取其150 μL和200 μL的Bm25.8 (OD600值达1.1-1.4)细菌培养物混合,31℃静置30 min,再加入400 μL LB液体培养基,然后置于摇床上于31℃,225 rpm下培养1 h,之后取5μL感染的细菌培养物涂布到LB/Carb平板上,37℃下培养待长出单克隆,取细菌单克隆于LB液体培养基中培养,送invitrogen公司测序。
测序获得的序列剔除载体序列、引物序列和PolyA后转换成FASTA格式文件输出,利用contigexpress软件进行序列的拼接,参数为程序默认值,拼接后得到一致序列,包括由多条ESTs拼接成的跨叠群(contigs)及单一序列(singleton)。用Blastn和Blastx对所得序列进行比对、查询和注释。
作者贡献
栗婷和朱彩虹是本研究的实验设计和实验研究的执行人;栗婷完成数据分析,论文初稿的写作;齐力旺参与实验设计,试验结果分析;韩素英是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究由国家“973”项目(2009CB119100),国家自然科学基金重点项目(30830086),国家“863”项目(SQ2010AA1000687004,2006AA100109, 2008AA10Z126)和国家林业局948项目(2007-4-03)资助。
参考文献
Cairly J., and Pullman G.S., 2007, The cellular and molecular biology of conifer embryogenesis, New Phytologist, 176(3): 511-536
Cuming A.C., and Lane B.G., 1979, Protein synthesis in imbibing wheat embryos, European Journal of Biochemistry, 99(2): 217-224
Davidonis G.H., and Hamilton R.H., 1983, Plant regeneration from callus of Gossypium hirsulum L., Plant Science Letters, 32(1-2): 89-93
Filonova L.H., Bozhkov P.V., and Arnold S.V., 2000, Development pathway of somatic embryogenesis in Picea abies as revealed by time lapse tracking,Journal of Experimental Botany, 51(343): 249-264
Ikeda-Iwai M., Umehara M., Satoh S., and Kamada H., 2003, Stress-induced somatic embryogenesis in vegetative tissues of Arabidopsis thaliana, The Plant Journal, 34(1): 107-114
Litts J.C., Erdman M.B., Huang N., Karrer E.E., Noueiry A., Quatrano R.S., and Rodriguez R.L., 1992, The nucleotide sequence of the rice(Oryza sativa) Em protein gene (EMP1), Plant Molecular Biology, 19(2): 335-337
Long S.H., Chen X.B., Deng X., and Gao Y., 2008,Construction of Flax Full- length cDNA Library with SMART System, Zhongguo Maye Kexue (Plant Fiber Sciences in China), 30(3): 128-140 (龙松华, 陈信波, 邓欣, 高原, 2008, SMART技术构建亚麻韧皮部全长cDNA文库, 中国麻业科学, 30(3): 128-140)
Lv S.F., Zhang S.G., Qi L.W., Sun X.M., and Wang J.H., 2003, Application of Somatic Embryognenesis in Woody Species, Shijie Linye Yanjiu (World Forestry Research), 16(6): 6-10 (吕守芳, 张守攻, 齐力旺, 孙晓梅, 王建华, 2003, 体细胞胚胎发生在林木树种中的应用, 世界林业研究, 16(6): 6-10)
Lv S.F., Zhang S.G., Qi L.W., Sun X.M., and Wang J.H., 2005, Somatic Embryogenesis from Immature Embryos of Larix kaempferi, Linye Kexue (Scientia Silvae Sinicae), 41(2): 48-53 (吕守芳, 张守攻, 齐力旺, 孙晓梅, 王建华, 2005, 日本落叶松体细胞胚胎发生的研究, 林业科学, 2005, 41(2): 48-53)
Mao X.G., Jing R.L., Kong X.Y., Zhao G.Y., and Jia J.Z., 2006, Comparison of Methods to Construct a Ful1-Length CDNA Library, Yichuan (Hereditas), 28(7): 865-873 (毛新国, 景蕊莲, 孔秀英, 赵光耀, 贾继增, 2006, 几种全长cDNA文库构建方法比较, 遗传, 28(7): 865-873)
Palovaara J., Hallberg H., Stasolla C., Luit B., and Hakman I., 2010, Expression of a gymnosperm PIN homologous gene correlates with auxin immunolocalization pattern at cotyledon formation and in demarcation of the procambium during Picea abies somatic embryo development and in seedling tissues, Tree Physiology, 30(4): 479-489
Pullman G.S., Johnson S., Peter G., Cairney J., and Xu N., 2003, Improving loblolly pine somatic embryo maturation:comparison of somatic and zygotic embryo morphology, germination, and gene expression, Plant Cell Reports, 21(8): 747-758
Ralph S.G., Chun H.J.E., Kolosova1 N., Cooper D., Oddy C., Ritland C.E., Kirkpatrick R., Moore R., Barber S., Holt R.A., Jones S.J., Marra M.A., Douglas C.J., Ritland K., and Bohlmann J., 2008, A conifer genomics resource of 200,000 spruce (Picea spp.) ESTs and 6,464 high-quality, sequence-finished full-length cDNAs for Sitka spruce (Picea sitchensis), BMC Geonomics, 9: 484
Steward F.C., Mapes M.O., and Mears K., 1958, Growth and organized development of cultured cells.II.Organization in cultures grown from freely suspended cells, Am. J. Bot, 45: 653-704
Wang B., Ai X.L., Wang Z.F., Li F., and Luo M., 2008, cDNA library Construction and EST Analysis of Saussurea inmlucrata Kar. et Kit, Xinjiang Nongye Kexue(Xinjiang Agricultural Sciences), 45(1): 56-60 (王博, 艾秀莲, 王志方, 李芳, 罗明, 2008, 新疆雪莲全长eDNA文库构建及表达序列标签(ESTs)特性分析, 新疆农业科学, 45(1): 56-60)
Wang P., Wang G., Ji J., Zeng F.T., Huang B.C., Cao J., and Wu Y., 2004, Studies of Somatic Embryogenesis and Genetic Transformation by Agrobacterium, mediated in Soybean, Hereditas, 26(5): 695-700 (王萍, 王罡, 季静, 曾凡亭, 黄彬城, 曹江, 吴颖, 2004, 大豆体细胞胚胎发生与农杆菌介导的遗传转化, 遗传, 2004, 26(5): 695-700)
Williams B., and Tsang A., 1991, A maize gene expressed during embryogenesis is abscisic-acid inducible and highly conserved, Plant Molecular Biology, 16(5): 919-923
Zhang L., Han S.Y., Zhang S.G., and Qi L.W., 2006, Advances in Research in Plant Somatic Embryogenesis and the Relational Responding Genes, Biotechnology Bulletin, supplement: 1-5 (张蕾, 韩素英, 张守攻, 齐力旺, 2006, 植物体细胞胚胎发生及其相关应答基因的研究进展, 生物技术通报, 增刊: 1-5)
Zhang L., Qi L.W., Han S.Y., 2009, Diferentially expressed genes during Larix somatic embryo maturation and the expression profile of partial genes, Yichuan (Hereditas), 31(5): 540-545 (张蕾, 齐力旺, 韩素英, 2009, 落叶松体细胞胚成熟阶段差异表达的基因及部分基因的表达谱分析, 遗传, 31(5): 540-545)
Zhou X.M., Zhang B.Y., Su X..H., Wang D.H., Huang Q.J., Zhang X.H., and Zhang Z.Y., 2007, Construction of Full-length cDNA Library of Male Floral Buds of Populus deltoide, Forest Research, 20(4): 469-472(周祥明, 张冰玉, 苏晓华, 王大海, 黄秦军, 张香华, 张志毅, 2007, 美洲黑杨雄性花芽全长cDNA文库构建, 林业科学研究, 20(4): 469-472)
Zhu Y.Y., Machleder E.M., Chenchik A., Li R., and Siebert P.D., 2001, Reverse transcriptase template switching: a SMART approach for full-length cDNA library construction, Bio. Techniques, 30(4): 892-897